Page 209 - 《广西植物》2023年第5期
P. 209
9 7 6 广 西 植 物 43 卷
表 1 云南樱花品种‘红霞’的叶绿体基因组 b / f 复 合 物 ( petB、 petL、 petN、 petG、 petD、 petA )、
Table 1 Chloroplast genome of flowering cheery RubisCO 大亚基( rbcL)、成熟酶( matK)、光系统 I
cultivar Cerasus‘Hongxia’ in Yunnan
(psaI、psaA、psaB、psaJ、psaC)、核糖体蛋白( SSU)
鸟嘌呤和胞嘧啶
起点 终点 长度 ( rps2、 rps11、 rps3、 rps8、 rps16、 rps19、 rps15、 rps4、
区域 Guanine and
Start End Length
Region Cytosine rps12、rps14、 rps7、 rps18)、 光 系 统 Ⅱ ( psbM、 psbH、
(bp) (bp) (bp)
(%)
psbI、psbE、psbC、psbJ、psbZ、psbL、psbK、psbA、psbN、
全基因组 1 157 832 157 832 36.73
psbB、psbT、 psbD、 psbF)、 其 他 基 因 ( accD、 cemA、
Chloroplast genome
大单拷贝区 1 85 950 85 950 34.61 ccsA)ꎮ
LSC
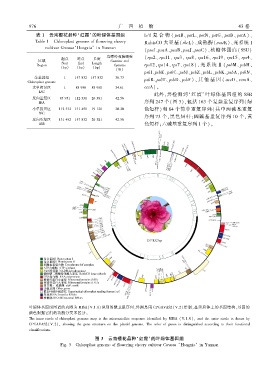
此外ꎬ共检测到‘ 红霞’ 叶绿体基因组的 SSR
反向重复区 85 951 112 331 26 381 42.56
IRA 序列 247 个(图 3)ꎬ包括 163 个复杂重复序列( 绿
小单拷贝区 112 332 131 451 19 120 30.20 色短杆)和 84 个简单重复序列( 其中两碱基重复
SSC
序列 73 个ꎬ黑色短杆ꎻ四碱基重复序列 10 个ꎬ黄
反向重复区 131 452 157 832 26 381 42.56
IRB 色短杆ꎻ六碱基重复序列 1 个)ꎮ
叶绿体基因组图谱的内圈为 MISA(V.1.0)识别的微卫星序列ꎬ外圈是用 CPGAVAS2(V.2)绘制ꎬ显示质体上的基因结构ꎬ基因的
颜色根据它们的功能分类来区分ꎮ
The inner circle of chloroplast genome map is the microsatellite sequence identified by MISA (V.1.0)ꎬ and the outer circle is drawn by
CPGAVAS2( V. 2)ꎬ showing the gene structure on the plastid genome. The color of genes is distinguished according to their functional
classifications.
图 3 云南樱花品种‘红霞’的叶绿体基因组
Fig. 3 Chloroplast genome of flowering cheery cultivar Cerasus ‘Hongxia’ in Yunnan