Page 57 - 《广西植物》2020年第9期
P. 57
1 2 7 2 广 西 植 物 40 卷
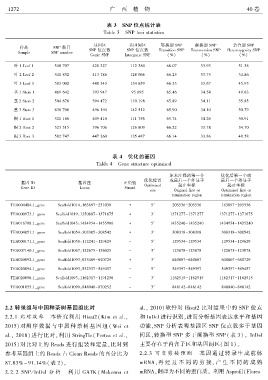
表 3 SNP 位点统计表
Table 3 SNP loci statistics
基因区 基因间区 转换型 SNP 颠换型 SNP 杂合型 SNP
样品 SNP 数目
SNP 位点数 SNP 位点数 Transition SNP Transversion SNP Heterozygosity SNP
Sample SNP number
Genic SNP Intergenic SNP (%) (%) (%)
叶 1 Leaf 1 540 707 428 327 112 380 66.07 33.93 51.38
叶 2 Leaf 2 541 852 413 786 128 066 66.25 33.75 34.86
叶 3 Leaf 3 583 002 448 343 134 659 66.33 33.67 45.95
茎 1 Stem 1 489 642 393 947 95 695 65.46 34.54 49.63
茎 2 Stem 2 504 670 394 472 110 198 65.89 34.11 35.85
茎 3 Stem 3 638 706 496 194 142 512 65.90 34.10 50.79
根 1 Root 1 521 166 409 410 111 756 65.74 34.26 50.91
根 2 Root 2 523 515 396 706 126 809 66.22 33.78 34.70
根 3 Root 3 582 747 447 260 135 487 66.14 33.86 48.58
表 4 优化的基因
Table 4 Gene structure optimized
原来注释的第一个 优化后第一个或
优化位置 或最后一个外显子 最后一个外显子
基因 ID 基因座 正负链
Optimized 起止坐标 起止坐标
Gene ID Locus Strand
site Original first or Optimized first or
termination region termination region
TEA004484.1_gene Scaffold1014:163897-231030 + 5′ 205536-205536 163897-205536
TEA000973.1_gene Scaffold1019:1350887-1371675 + 3′ 1371277-1371277 1371277-1371675
TEA016708.1_gene Scaffold1045:1434954-1455906 + 5′ 1435240-1435240 1434954-1435240
TEA004051.1_gene Scaffold1054:303905-308542 + 3′ 308318-308318 308318-308542
TEA000171.1_gene Scaffold1058:112241-129629 - 5′ 129534-129534 129534-129629
TEA007140.1_gene Scaffold1067:122675-156025 - 3′ 123878-123878 122675-123878
TEA020092.1_gene Scaffold1093:635489-640729 + 3′ 640607-640607 640607-640729
TEA020094.1_gene Scaffold1093:532337-549457 - 5′ 549397-549397 549397-549457
TEA020098.1_gene Scaffold1093:1162317-1191258 - 3′ 1162515-1162515 1162317-1162515
TEA001055.1_gene Scaffold1099:848040-870252 - 3′ 848142-848142 848040-848142
2.2 转录组与中国种茶树基因组比对 al.ꎬ 2010)软件对 Hisat2 比对结果中的 SNP 位点
2.2.1 比对效率 本研究利用 Hisat2( Kim et al.ꎬ 和 InDel 进行识别ꎬ进而分析基因表达水平和基因
2015)对测 序 数 据 与 中 国 种 茶 树 基 因 组 ( Wei et 功能ꎬSNP 分析表明基因区 SNP 位点数多于基因
al.ꎬ 2018) 进行比对ꎬ利用 StringTie( Pertea et al.ꎬ 间区ꎬ转换型 SNP 多于颠换型 SNP( 表 3)ꎮ InDel
2015)对比对上的 Reads 进行组装和定量ꎬ比对到 主要存在于内含子区和基因间区(图 1)ꎮ
参考基因组上的 Reads 占 Clean Reads 的百分比为 2.2.3 可 变 剪 接 预 测 基 因 通 过 转 录 生 成 前 体
mRNAꎬ再 经 过 不 同 的 剪 接ꎬ 产 生 不 同 的 成 熟
87.83% ~ 91.14%(表 2)ꎮ
2.2. 2 SNP / InDel 分 析 利 用 GATK ( Mckenna et mRNAꎬ翻译为不同的蛋白质ꎮ 利用 Asprofil(Florea