Page 76 - 《广西植物》2020年第9期
P. 76
9 期 涂冬萍等: 牛大力淀粉酶基因家族的生物信息学分析 1 2 9 1
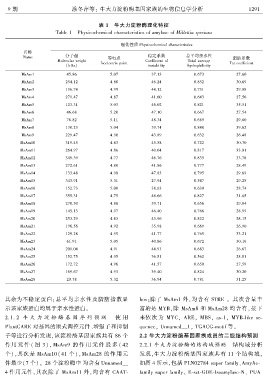
表 1 牛大力淀粉酶理化特征
Table 1 Physicochemical characteristics of amylase of Millettia speciosa
理化性质 Physicochemical characteristics
名称
Name 分子量 等电点 稳定系数 总平均亲水性 脂肪系数
Molecular weight Coefficient of Total average
Isoelectric point Fat coefficient
(kDa) instability hydrophilicity
MsAm1 45.86 5.07 37.13 0.673 27.60
MsAm2 254.12 4.80 46.24 0.852 30.69
MsAm3 136.74 4.99 44.12 0.731 29.08
MsAm4 271.47 4.87 41.60 0.643 27.56
MsAm5 123.31 5.03 46.02 0.821 35.51
MsAm6 46.68 5.20 47.10 0.667 27.54
MsAm7 78.82 5.11 48.34 0.689 29.60
MsAm8 130.23 5.04 39.74 0.848 39.62
MsAm9 229.47 4.90 43.89 0.632 26.48
MsAm10 319.45 4.83 45.58 0.722 30.70
MsAm11 264.97 4.86 40.04 0.817 33.01
MsAm12 349.39 4.77 46.76 0.835 33.78
MsAm13 272.61 4.80 41.86 0.777 28.49
MsAm14 133.48 4.98 47.83 0.795 29.69
MsAm15 343.91 5.31 27.94 0.547 29.25
MsAm16 152.73 5.00 38.63 0.630 28.74
MsAm17 355.31 4.75 46.66 0.827 31.65
MsAm18 270.30 4.88 39.71 0.656 29.84
MsAm19 145.13 4.97 46.40 0.766 28.99
MsAm20 253.29 4.83 43.96 0.822 28.15
MsAm21 196.56 4.92 35.98 0.669 26.90
MsAm22 129.28 4.95 41.77 0.765 33.21
MsAm23 61.91 5.05 40.86 0.672 30.16
MsAm24 201.04 4.91 44.93 0.683 26.67
MsAm25 152.75 4.95 36.51 0.562 28.81
MsAm26 172.72 4.96 41.57 0.650 27.59
MsAm27 169.67 4.93 39.40 0.824 30.20
MsAm28 20.78 5.32 36.94 0.781 31.25
其余为不稳定蛋白ꎻ总平均亲水性及脂肪指数显 boxꎻ除了 MsAm1 外ꎬ均含有 STRE ꎮ 其次含量丰
示该家族蛋白均属于亲水性蛋白ꎮ 富的是 MYBꎬ除 MsAm8 和 MsAm28 均含有ꎬ接下
2.1. 2 牛 大 力 淀 粉 酶 基 因 序 列 预 测 使 用 来依 次 为 MYCꎬ AREꎬ MBSꎬ as ̄1ꎬ MYB ̄like se ̄
PlantCARE 对基因的顺式调控元件、增强子和抑制 quenceꎬ Unnamed__1ꎬ TGACG ̄motif 等ꎮ
子等进行分析发现ꎬ该淀粉酶基因家族共有 86 个 2.2 牛大力淀粉酶基因家族成员的二级结构预测
作用 元 件 ( 图 3)ꎬ MsAm9 的 作 用 元 件 最 多 ( 42 2.2.1 牛大力淀粉酶的结构域预测 结构域分析
个)ꎬ其次是 MsAm10(41 个)ꎬMsAm28 的作用元 发现ꎬ牛大力淀粉酶基因家族共有 11 个结构域ꎬ
件最少(7 个)ꎮ 28 个淀粉酶中 均含有 Unnamed__ 如图 4 所示ꎬ包括 PLN02784 super family、AmyAc ̄
4 作用元件ꎬ其次除了 MsAm11 外ꎬ均含有 CAAT ̄ family super family、 E ̄set ̄GDE ̄Isoamylase ̄N、 PUA