Page 56 - 《广西植物》2020年第1期
P. 56
5 2 广 西 植 物 40 卷
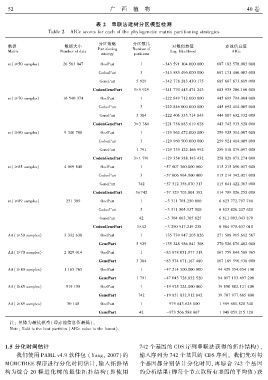
表 2 串联法建树分区模型检测
Table 2 AICc scores for each of the phylogenetic matrix partitioning strategies
分区策略 分区数目
数据 数据大小 对数似然值 赤池信息值
Partitioning Number of
Matrix Number of data Log-likelihood AICc
strategy partitions
nt(≥50 samples) 26 563 047 OnePart 1 -343 591 104.000 000 687 182 578.003 000
CodonPart 3 -343 585 496.000 000 687 171 406.003 000
GenePart 5 929 -342 778 283.430 175 685 687 673.619 000
CodonGenePart 3×5 929 -341 770 443.474 243 683 935 206.166 000
nt(≥70 samples) 16 540 374 OnePart 1 -222 849 712.000 000 445 699 794.004 000
CodonPart 3 -222 846 000.000 000 445 692 414.005 000
GenePart 3 384 -222 406 333.714 843 444 887 632.932 000
CodonGenePart 3×3 384 -221 758 883.619 628 443 742 935.528 000
nt(≥80 samples) 9 340 788 OnePart 1 -129 962 472.000 000 259 925 314.007 000
CodonPart 3 -129 960 500.000 000 259 921 414.009 000
GenePart 1 791 -129 739 122.166 992 259 518 079.097 000
CodonGenePart 3×1 791 -129 354 388.143 432 258 828 073.274 000
nt(≥85 samples) 4 069 848 OnePart 1 -57 607 360.000 000 115 215 090.017 000
CodonPart 3 -57 606 964.000 000 115 214 342.021 000
GenePart 742 -57 512 358.070 313 115 041 422.363 000
CodonGenePart 3×742 -57 329 703.801 392 114 709 026.252 000
nt(≥89 samples) 231 309 OnePart 1 -3 311 701.250 000 6 623 772.797 760
CodonPart 3 -3 311 505.937 500 6 623 426.247 620
GenePart 42 -3 304 863.765 625 6 611 003.043 870
CodonGenePart 3×42 -3 290 917.219 238 6 584 975.637 010
AA(≥50 samples) 3 332 638 OnePart 1 -135 739 947.205 826 271 508 915.612 567
GenePart 5 929 -135 248 986.841 308 270 526 876.482 000
AA(≥70 samples) 2 029 014 OnePart 1 -83 878 051.077 318 167 759 844.588 965
GenePart 3 384 -83 574 671.167 480 167 169 596.938 000
AA(≥80 samples) 1 165 765 OnePart 1 -47 214 500.000 000 94 429 354.054 100
GenePart 1 791 -47 043 728.832 520 94 097 113.497 200
AA(≥85 samples) 519 158 OnePart 1 -19 925 224.000 000 39 850 802.121 400
GenePart 742 -19 851 832.912 842 39 707 977.665 800
AA(≥89 samples) 30 148 OnePart 1 -979 643.625 000 1 959 681.828 340
GenePart 42 -973 566.588 867 1 948 059.215 120
注: 黑体为最优模型(即赤池信息值最低)ꎮ
Note: Bold is the best partition (AICc value is the lowest).
1.5 分化时间估计 742 个基因的 CDS 序列串联法获得的拓扑结构)ꎬ
我们使用 PAML v4.9 软件包 (Yangꎬ 2007)的 输入序列为 742 个基因的 CDS 序列ꎮ 我们先对每
MCMCTREE 程序进行分化时间估计ꎬ输入拓扑结 个基因都分别估计分化时间ꎬ再综合 742 个基因
构为综合 20 棵进化树的最佳拓扑结构( 即使用 的分析结果(即每个节点取所有基因的平均值) 获