Page 107 - 《广西植物》2025年第12期
P. 107
12 期 雷翰霖等: 锦葵科 Malvatheca 分支的质体基因组演化及系统发育分析 2 2 4 9
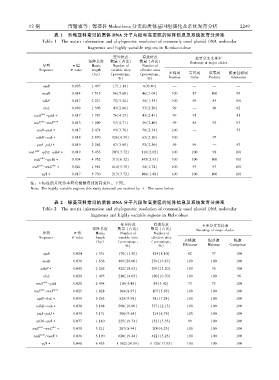
表 1 木棉亚科常见的质体 DNA 分子片段和高变区的矩阵信息及系统发育分辨率
Table 1 The matrix information and phylogenetic resolution of commonly used plastid DNA molecular
fragments and highly variable regions in Bombacoideae
变异位点 有效位点 主要分支支持率
矩阵长度 数量 (占比) 数量 (占比) Bootstrap of major clades
序列 π 值 Matrix Number of Number of
Sequence π value length variable sites effective sites
(bp) (percentageꎬ (percentageꎬ 木棉属 吉贝属 瓜栗属 猴面包树属
Bombax Ceiba Pachira Adansonia
%) %)
atpB 0.003 1 497 17(1.14) 6(0.40) — — — —
matK 0.014 1 515 86(5.68) 46(3.04) 100 87 100 99
ndhF 0.017 2 221 72(3.24) 34(1.53) 100 99 83 100
rbcL 0.009 1 503 43(2.86) 33(2.20) 99 — 66 62
trnH GUG -psbA∗ 0.017 1 781 76(4.27) 43(2.41) 99 95 — 81
trnL UAA -trnF GAA 0.013 1 209 57(4.71) 29(2.40) 99 89 92 97
accD-psaI∗ 0.017 2 474 93(3.76) 58(2.34) 100 — 53
ndhD-ccsA∗ 0.018 2 870 126(4.39) 63(2.20) 100 — 97 —
petA-psbJ∗ 0.019 2 262 87(3.85) 52(2.30) 99 99 — 97
trnL UAG -rpl32-ndhF∗ 0.019 5 455 203(3.72) 110(2.02) 100 100 98 100
trnK UUU -rps16∗ 0.034 4 952 313(6.32) 145(2.93) 100 100 100 100
trnS GCU -trnG UCC ∗ 0.021 1 981 114(5.75) 34(1.72) 100 97 97 100
ycf1∗ 0.017 5 730 213(3.72) 108(1.88) 100 100 100 100
注: ∗标注的片段为本研究检测得到的高变区ꎮ 下同ꎮ
Note: The highly variable regions this study detected are marked by ∗. The same below.
表 2 锦葵亚科常见的质体 DNA 分子片段和高变区的矩阵信息及系统发育分辨率
Table 2 The matrix information and phylogenetic resolution of commonly used plastid DNA molecular
fragments and highly variable regions in Malvoideae
变异位点 有效位点 主要分支支持率
矩阵长度 数量 (占比) 数量 (占比) Bootstrap of major clades
序列 π 值 Matrix Number of Number of
Sequence π value length variable sites effective sites
(bp) (percentageꎬ (percentageꎬ 木槿族 锦葵族 棉族
%) %) Hibisceae Malveae Gossypieae
atpB 0.024 1 531 176(11.50) 124(8.10) 82 97 100
matK 0.036 1 636 409(25.00) 276(16.87) 100 100 100
ndhF∗ 0.040 2 265 422(18.63) 299(13.20) 100 95 100
rbcL 0.020 1 495 210(14.05) 160(10.70) 100 100 96
trnH GUG -psbA 0.020 2 454 110(4.48) 84(3.42) 73 73 100
trnL UAA -trnF GAA 0.025 1 828 164(8.97) 107(5.85) 100 100 100
atpB-rbcL∗ 0.039 5 263 525(9.98) 381(7.24) 100 100 100
ndhD-ccsA∗ 0.038 3 108 500(16.09) 377(12.13) 100 100 100
petA-psbJ∗ 0.039 3 171 306(9.65) 214(6.75) 100 100 100
rpl36-rps8∗ 0.037 1 140 225(19.74) 152(13.33) 99 100 100
trnS GCU -trnG UCC ∗ 0.038 3 211 287(8.94) 200(6.23) 100 100 100
trnK UUU / matK∗ 0.036 3 190 620(19.44) 422(13.23) 100 100 100
ycf1∗ 0.048 6 433 1 582(24.59) 1 128(17.53) 100 100 100