Page 79 - 《广西植物》2020年第9期
P. 79
1 2 9 4 广 西 植 物 40 卷
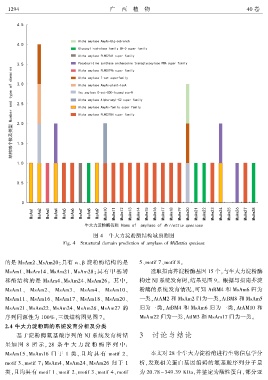
图 4 牛大力淀粉酶结构域预测图
Fig. 4 Structural domain prediction of amylase of Millettia speciosa
的是 MsAm2、MsAm20ꎻ具有 α、β 淀粉酶结构的是 5、motif 7、motif 8ꎮ
MsAm1、MsAm14、MsAm21、MsAm28ꎻ具 有 甲 基 转 选取拟南芥淀粉酶基因 15 个ꎬ与牛大力淀粉酶
移酶结构的是 MsAm4、MsAm24、MsAm26ꎮ 其中ꎬ 构建 NJ 系统发育树ꎬ结果见图 9ꎮ 根据与拟南芥淀
MsAm1、 MsAm2、 MsAm3、 MsAm4、 MsAm10、 粉酶的系统发育情况ꎬ可知 AtBM4 和 MsAm6 归为
MsAm11、 MsAm16、 MsAm17、 MsAm18、 MsAm20、 一类ꎬAtAM2 和 MsAm2 归为一类ꎬAtBM8 和 MsAm5
MsAm21、MsAm22、 MsAm24、 MsAm26、 MsAm27 的 归为一类ꎬAtBM4 和 MsAm6 归为一类ꎬAtAM10 和
序列同源性为 100%ꎬ三级结构图见图 7ꎮ MsAm22 归为一类ꎬAtIM3 和 MsAm17 归为一类ꎮ
2.4 牛大力淀粉酶的系统发育分析及分类
基于淀粉酶氨基酸序列的 NJ 系统发育树结 3 讨论与结论
果如 图 8 所 示ꎬ 28 条 牛 大 力 淀 粉 酶 序 列 中:
MsAm15、MsAm16 归 于 1 类ꎬ 且 均 具 有 motif 2、 本文对 28 个牛大力淀粉酶进行生物信息学分
motif 3、motif 7ꎻMsAm4、MsAm24、MsAm26 归 于 1 析ꎬ发现相关蛋白基因编码的氨基酸序列分子量
类ꎬ且均具有 motif 1、motif 2、motif 3、motif 4、motif 为 20.78 ~ 349.39 KDaꎬ并鉴定为酸性蛋白ꎬ部分亚