Page 81 - 《广西植物》2025年第7期
P. 81
7 期 何斌等: 刺梨全长转录组测序分析及黄酮类化合物生物合成相关基因挖掘 1 2 7 3
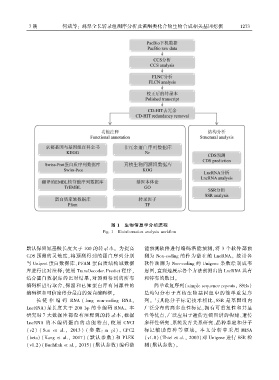
图 1 生物信息学分析流程
Fig. 1 Bioinformatics analysis workflow
默认保留氨基酸长度大于 100 的转录本ꎮ 为提高 能预测软件进行编码潜能预测ꎬ将 3 个软件都预
CDS 预测的灵敏度ꎬ将预测得到的蛋白序列分别 测为 Non ̄coding 的作为潜在的 LncRNAꎮ 统计各
与 Uniprot 蛋白数据库、PFAM 蛋白质结构域数据 软件预测为 Non ̄coding 的 Unigene 条数绘制成韦
库进行比对注释ꎻ使用 TransDecoder.Predict 程序ꎬ 恩图ꎬ直观地展示各个方法预测出的 LncRNA 共有
结合蛋白数据库的比对结果ꎬ对预测得到的所有 和特有的数目ꎮ
编码框进行取舍ꎬ保留和已知蛋白库有同源性的 简单重复序列(simple sequence repeatsꎬ SSRs)
编码框和可信度得分最高的蛋白编码框ꎮ 是均匀分布于真核生物基因组中的简单重复序
长 链 非 编 码 RNA ( long non ̄coding RNAꎬ 列ꎮ 与其他分子标记技术相比ꎬSSR 是基因组内
LncRNA)是长度大于 200 bp 的非编码 RNAꎮ 本 广泛分布的高多态性标记ꎬ拥有可重复性和共显
研究对 7 大数据库都没有注释到的转录本ꎬ根据 性等优点ꎬ广泛应用于遗传连锁图谱的构建、遗传
LncRNA 的 不 编 码 蛋 白 的 功 能 特 点ꎬ 使 用 CNCI 多样性研究、系统发育关系研究、品种鉴定和分子
(v2 ) ( Sun et al.ꎬ 2013 ) ( 参 数: m pl )、 CPC2 标记 辅 助 育 种 等 领 域ꎮ 本 文 分 析 中 采 用 MISA
(beta) ( Kang et al.ꎬ 2017) ( 默 认 参 数) 和 PLEK (v1.0)(Thiel et al.ꎬ 2003)对 Unigene 进行 SSR 检
(v1.2)(Buchfink et al.ꎬ 2015) (默认参数) 编码潜 测(默认参数)ꎮ