Page 82 - 《广西植物》2025年第7期
P. 82
1 2 7 4 广 西 植 物 45 卷
921 786 799 bpꎬ数目为 351 714 条ꎬ平均长度为
2 结果与分析 2 621 bpꎬN50 长 度 为 2 759 bpꎮ 其 中ꎬ FLNC 为
316 269条ꎬ数据量为 803 609 064 bpꎬ平均长度为
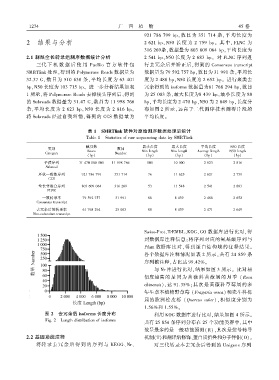
2.1 刺梨全长转录组测序数据统计分析 2 541 bpꎬN50 长度为 2 683 bpꎮ 对 FLNC 序列进
三代 下 机 数 据 后 使 用 PacBio 官 方 软 件 包 行去冗余后并矫正后ꎬ得到的 Consensus transcript
SMRTlink 处理ꎬ得到的 Polymerase Reads 数据量为 数据量为 79 592 757 bpꎬ数目为 31 991 条ꎬ平均长
32.37 Gꎬ数目为 510 638 条ꎬ平均长度为 63 401 度为 2 488 bpꎬN50 长度为 2 652 bpꎮ 进行聚类去
bpꎬN50 长度为 103 715 bpꎮ 进一步分析结果如表 冗余得到的 isoforms 数据量为61 768 294 bpꎬ数目
1 所示ꎬ将 Polymerase Reads 去掉接头序列后ꎬ得到 为 25 003 条ꎬ最大长度为8 439 bpꎬ最小长度为 88
的 Subreads 数据量为 31.47 Gꎬ数目为 11 998 766 bpꎬ平均长度为 2 470 bpꎬN50 为 2 649 bpꎬ长度分
条ꎬ平均长度为 2 623 bpꎬN50 长度为 2 816 bpꎮ 布如图 2 所示ꎬ远高于二代测序技术测得片段的
将 Subreads 经过自我纠错ꎬ得到的 CCS 数据量为 平均长度ꎮ
表 1 SMRTlink 软件对原始测序数据处理后统计
Table 1 Statistics of raw sequencing data by SMRTlink
碱基数 最小长度 最大长度 平均长度 N50 长度
类别 数目
Bases Min length Max length Average length N50 length
Category Number
(bp) (bp) (bp) (bp) (bp)
子读序列 31 470 000 000 11 998 766 100 10 000 2 623 2 816
Subread
环状一致性序列 921 786 799 351 714 76 11 625 2 621 2 759
CCS
全长非嵌合序列 803 609 064 316 269 53 11 548 2 541 2 683
FLNC
一致转录本 79 592 757 31 991 88 8 439 2 488 2 652
Consensus transcript
去冗余后的转录本 61 768 294 25 003 88 8 439 2 471 2 649
Non ̄redundant transcript
Swiss ̄Prot、TrEMBL、KOG、GO 数据库进行比对ꎬ得
到数据库注释信息ꎻ将序列对应的氨基酸序列与
Pfam 数据库比对ꎬ得到蛋白结构域的注释结果ꎮ
各个数据库注释情况如表 2 所示ꎬ共有 24 859 条
序列被注释ꎬ占比达 99.42%ꎮ
与 Nr 库进行比对ꎬ结果如图 3 所示ꎮ 比对相
似度最高 的 是 同 为 蔷 薇 科 蔷 薇 属 的 月 季 ( Rosa
chinensis)ꎬ达 91.35%ꎻ其次是蔷薇科草莓属的多
年生草本植物野草莓 (Fragaria vesca) 和壳斗科栎
属的欧洲栓皮栎 ( Quercus suber)ꎬ相似度分别为
1.56%和 1.55%ꎮ
图 2 去冗余后 isoforms 长度分布 利用 KOG 数据库进行比对ꎬ结果如图 4 所示ꎮ
Fig. 2 Length distribution of isoforms 共有 25 854 条序列分布在 25 个功能类群中ꎬ其中
数量最多的是一般功能预测(R)ꎬ其次是信号转导
2.2 基因功能注释 机制(T)和翻译后修饰、蛋白质折叠和分子伴侣(O)ꎮ
将转 录 去 冗 余 后 得 到 的 序 列 与 KEGG、 Nr、 对三代转录本去冗余后得到的 Unigene 序列