Page 58 - 《广西植物》2020年第9期
P. 58
9 期 王芬等: 基于高通量测序的都匀地区福鼎大白种茶树根茎叶分析 1 2 7 3
表 5 新基因功能注释结果
Table 5 Results of functional annotation of new genes
注释数据库 新基因数目
Annotated databases New gene number
COG 2 363
GO 6 218
KEGG 3 948
KOG 5 833
Pfam 5 383
Swiss ̄Prot 6 900
eggNOG 9 243
NR 10 107
总和 Total 10 244
et al.ꎬ 2013) 软件对可变剪接类型和表达量进行
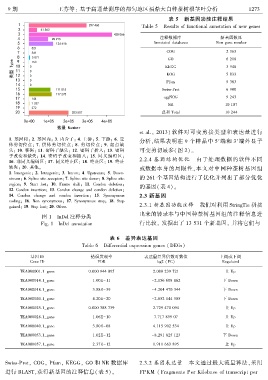
1. 基因间ꎻ 2. 基因内ꎻ 3. 内含子ꎻ 4. 上游ꎻ 5. 下游ꎻ 6. 受 分析ꎬ结果表明在 9 个样品中 5′端和 3′端外显子
体剪切位点ꎻ 7. 供体剪切位点ꎻ 8. 剪切位点ꎻ 9. 起点缺
失ꎻ 10. 移码ꎻ 11. 密码子缺失ꎻ 12. 密码子插入ꎻ 13. 密码 可变剪切最多(图 2)ꎮ
子改变和缺失ꎻ 14. 密码子改变和插入ꎻ 15. 同义编码区ꎻ 2.2.4 基因结构优化 由于处理数据的软件不同
16. 非同义编码区ꎻ 17. 同义终止区ꎻ 18. 终止区ꎻ 19. 终止
缺失ꎻ 20. 其他ꎮ 或数据本身的局限性ꎬ本文对中国种茶树基因组
1. Intergenicꎻ 2. Intragenicꎻ 3. Intronꎻ 4. Upstreamꎻ 5. Down ̄
的 261 个基因结构进行了优化并列出了部分优化
streamꎻ 6. Splice site acceptorꎻ 7. Splice site donorꎻ 8. Splice site
regionꎻ 9. Start lostꎻ 10. Frame shiftꎻ 11. Condon deletionꎻ
的基因(表 4)ꎮ
12. Condon insertionꎻ 13. Condon change and condon deletionꎻ
14. Condon change and condon insertionꎻ 15. Synonymous 2.3 新基因
codingꎻ 16. Non synonymousꎻ 17. Synonymous stopꎻ 18. Stop
gainedꎻ 19. Stop lostꎻ 20. Other. 2.3.1 新基因功能注释 我们对利用 StringTie 拼接
出来的转录本与中国种茶树基因组的注释信息进
图 1 InDel 注释分类
Fig. 1 InDel annotation 行比较ꎬ 发掘出了 13 531 个新基因ꎬ 并将它们与
表 6 差异表达基因
Table 6 Differential expression genes (DEGs)
基因 ID 错误发现率 表达量差异倍数对数值 上调或下调
Gene ID FDR log2 (FC) Regulated
TEA000001.1_gene 0.000 944 095 2.088 239 721 上 Up
TEA000014.1_gene 1.00E-11 -2.156 608 862 下 Down
TEA000018.1_gene 3.88E-39 -4.204 470 344 下 Down
TEA000020.1_gene 4.20E-20 -2.852 144 505 下 Down
TEA000025.1_gene 0.000 388 739 2.729 470 094 上 Up
TEA000028.1_gene 1.06E-10 7.717 899 07 上 Up
TEA000040.1_gene 3.80E-08 4.115 902 534 上 Up
TEA000053.1_gene 1.02E-12 -8.291 825 123 下 Down
TEA000057.1_gene 2.37E-12 1.911 663 895 上 Up
Swiss ̄Protꎬ COGꎬ Pfamꎬ KEGGꎬ GO 和 NR 数据库 2.3.2 基因表达量 本文通过最大流量算法ꎬ采用
进行 BLASTꎬ获得新基因的注释信息(表 5)ꎮ FPKM ( Fragments Per Kilobase of transcript per