Page 55 - 《广西植物》2023年第11期
P. 55
11 期 李娟等: 桃叶珊瑚属植物的叶绿体基因组结构特征及系统发育分析 2 0 1 5
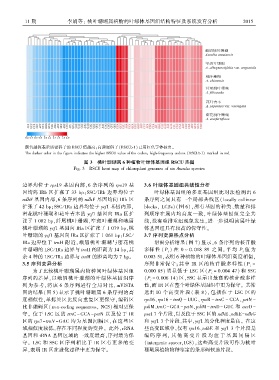
颜色越深表示该密码子的 RSCU 值越高ꎻ高频密码子(RSCU>1)已用红色字体标出ꎮ
The darker color in the figure indicates the higher RSCU value of the codonꎻ high ̄frequency codons (RSCU>1) marked in red.
图 3 桃叶珊瑚属 6 种植物叶绿体基因组 RSCU 热图
Fig. 3 RSCU heat map of chloroplast genomes of six Aucuba species
边界均位于 rps19 基因内部ꎬ6 条序列的 rps19 基 3.6 叶绿体基因组共线性分析
因均向 IRb 区扩张了 33 bpꎻSSC / IRb 边界均位于 叶绿体基因组的多重基因组比对法检测出 6
ndhF 基因内部ꎬ6 条序列的 ndhF 基因均向 IRb 区 条序列之间只有一个局部共线区( locally collinear
扩张了 42 bpꎻSSC / IRa 边界均位于 ycf1 基因内部ꎬ blocksꎬ LCBs)(图 6)ꎬ所有基因的种类、数量和排
密花桃叶珊瑚和花叶青木的 ycf1 基因向 IRa 区扩 列顺序在属内均高度一致ꎬ叶绿体基因组完全共
张了 1 082 bpꎬ纤尾桃叶珊瑚、窄斑叶珊瑚和峨眉 线ꎬ没有重排重组现象发生ꎬ进一步说明该属叶绿
桃叶珊瑚的 ycf1 基因向 IRa 区扩张了 1 079 bpꎬ桃 体基因组具有较高的保守性ꎮ
叶珊瑚的 ycf1 基因向 IRa 区扩张了 1 061 bpꎻLSC / 3.7 序列变异热点分析
IRa 边界位于 trnH 附近ꎬ峨眉桃叶珊瑚与密花桃 滑窗分析结果(图 7) 显示ꎬ6 条序列的核苷酸
叶珊瑚的 LSC / IRa 边界与 trnH 的距离为 14 bpꎬ其 多样 性 ( P ) 在 0 ~ 0. 018 89 之 间ꎬ 平 均 P 值 为
i
i
余 4 种的 LSC / IRa 边界与 trnH 的距离均为 7 bpꎮ 0.003 51ꎬ表明 6 种植物的叶绿体基因组高度相似ꎬ
3.5 序列变异分析 序列非常保守ꎻ其中 IR 区的核苷酸多样性( P =
i
为了比较桃叶珊瑚属内物种间叶绿体基因组 0.000 85) 明显低于 LSC 区(P = 0.004 47) 和 SSC
i
序列的差异ꎬ以峨眉桃叶珊瑚的叶绿体基因组序 (P = 0.006 14) 区ꎬSSC 显示出最高的核苷酸多样
i
列为参考ꎬ将该 6 条序列进行全局对比ꎬmVISTA 性ꎬ而 IR 区在整个叶绿体基因组中更为保守ꎮ 共筛
图的结果(图 5) 显示了桃叶珊瑚属 6 条序列的高 选出 10 个高变片段 ( 表 8)ꎬ 包括位于 LSC 区的
度相似性ꎬ单拷贝区比反向重复区更保守ꎬ编码区 rps16、rps16 - trnQ - UUG、 rpoB - trnC - GCA、 petN -
比非编码区(non ̄coding sequencesꎬ NCS)相对更保 psbM、trnC - GCA - petN、psbM - trnD - GUC 和 accD -
守ꎮ 位于 LSC 区的 trnC -GCA -petN 以及位于 IR psaI 7 个片段ꎬ以及位于 SSC 区的 ndhE、ndhE-ndhG
区的 rps7-trnV-GAC 均为基因间隔区ꎬ在这些区 和 ycf1 3 个片段ꎬ其中ꎬycf1 的分化程度最高ꎮ 在这
域相似度较低ꎬ存在不同程度的变异ꎮ 此外ꎬrRNA 些高变区域中ꎬ仅有 rps16、ndhE 和 ycf1 3 个片段是
基因和 tRNA 基因区域的一致度最高ꎬ序列最为保 编 码 序 列ꎬ 其 他 高 变 片 段 均 位 于 基 因 间 隔 区
守ꎮ LSC 和 SSC 区序列相比于 IR 区有更多的变 (intergenic spacerꎬIGS)ꎬ这些高变片段可作为桃叶
异ꎬ表明 IR 区在进化过程中更为保守ꎮ 珊瑚属植物物种鉴定的条形码候选片段ꎮ